
http://www.biologie.uni-hamburg.de/
lehre/bza/neuro/1ewk/dmglurem.htm

| 206 | 344 | 478 | 522 | 592 | 840 | ||||||||||||||
| extrazellulär | transmembran | intrazellulär | |||||||||||||||||
| ligandenbindend | Cys- reich |
||||||||||||||||||
| u | l | u | l | ||||||||||||||||
Die Ligandenbinderegion (LBR) wurde in einem Insekten-System exprimiert und das Proteinfragment kristallisiert. Nur dieser Teil des Rezeptors wird hier gezeigt. Eine intermolekulare Disulfidbrücke verbindet die Cysteine140 der beiden Ketten des Dimers miteinander. Die LBR kristallisierte als Dimer, aber die Abschnitte der Aminosäuren 125-153 sind in beiden Strängen ungeordnet und daher in der Röntgenstrukturanalyse nicht sichtbar
Das Bild hier zeigt das Protein parallel zur Membran gesehen - der nächste Knopf schaltet zu einer Ansicht entlang der Symmetrieachse des Dimers
Die Ligandenbinderegion ist deutlich in zwei Subdomänen unterteilt
In der anderen Proteinkette sind die Verhältnisse anders. Bei genauem Hinsehen bemerkt man eine Abweichung von der Symmetrie
Konsequenz: Es gibt zwei verschiedene stabile Konformationen in dem Homodimer
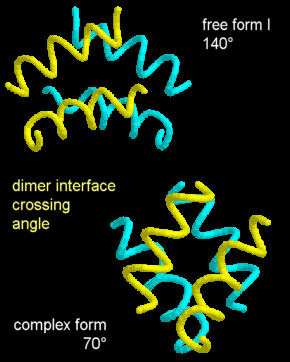
Bei Kristallisation des Proteins ohne Glutamat ergibt sich dieselbe Struktur, wobei die Glutamatbindestelle frei bleibt. Je nach verwendetem Puffersystem ergeben sich Strukturen der freien Form I oder II. In der freien Form I ist die Proteinkette wie in der Glutamat-gebundenen Form mit der weiten Furche gefaltet, aber die Lage der Monomeren unterscheidet sich bezüglich der Symmetrieachse.
Die Existenz verschiedener Konformere (unabhängig von Ligandenbindung und sogar innerhalb eines Homodimer-Kristalls) wird als dynamisches Gleichgewicht der Formen interpretiert. Der Übergang zwischen den Formen wird durch die ehr schwache Bindung der Helixpaare aneinander begünstigt. Die Bindung des Liganden führt sowohl zu einer Verschiebung des Gleichgewichtes als auch zu einer Stabilisierung des Komplexes mit der engen Furche (mit der festeren Bindung des Glutamates). Einen ähnlichen Mechanismus gibt es bei einem bakteriellen periplasmatischen Bindeprotein (LAOBP für Lysin/Arginin/Ornithin-Bindeprotein), das eine ähnliche Struktur aufweist.
Beide Domänen der LBR des Glutamat-Rezeptors haben eine alpha/beta-Topologie. In der der Zelloberfläche zugewandten Domäne
In der von der Zelloberfläche abgewandten Domäne
Das Protein ist nicht so "hohl" wie es in dieser Darstellung erscheint. Eine Überlagerung der Elektronenwolke gibt einen Eindruck des besetzten Volumens
Solange die Strukturdetails der übrigen Teile des Rezeptors noch nicht bekannt sind, können auch noch keine Aussagen zur Weitergabe der Signalerkennung durch die Glutamatbindung an das gekoppelte G-Protein gemacht werden.
Literatur:
Die Disulfidbrücke zwischen den Cys140 kann aufgrund der flexiblen Schlaufen ihrer Umgebung das Dimer nicht stabilisieren. Der Zusammenhalt der Dimere wird stattdessen durch Wechselwirkung zweier Helices-Paare bewirkt
Die gemischte beta-Strang-Topologie ist in diesem Schema wiedergegeben:

N Kunishima et al, Structural basis of glutamate regognition by a dimeric metabotropic glutamate receptor, Nature 407 (2000) 971-977
3-02 - Rolf Bergmann